壊れる瞬間から逆算する、分子の真実
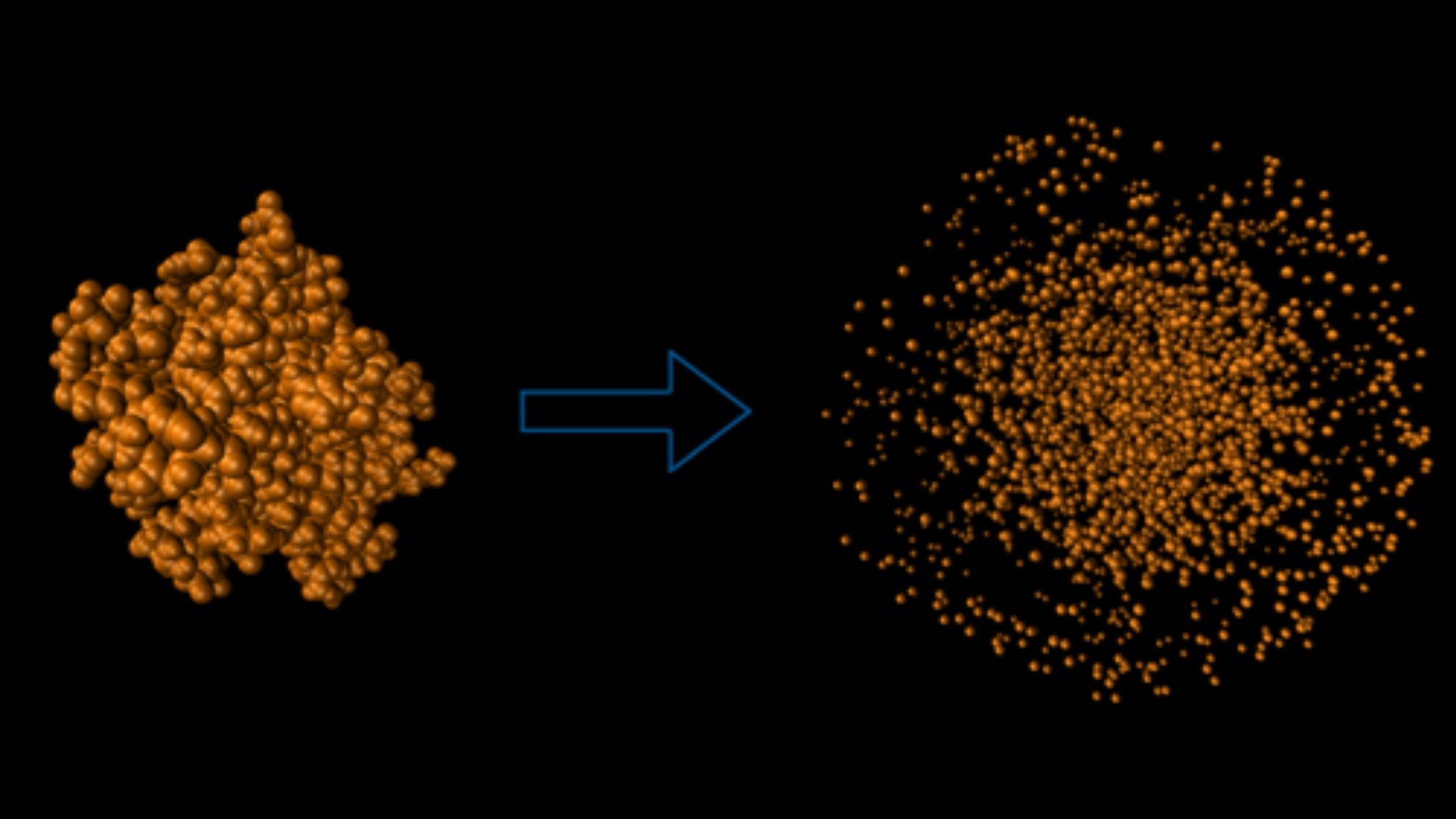
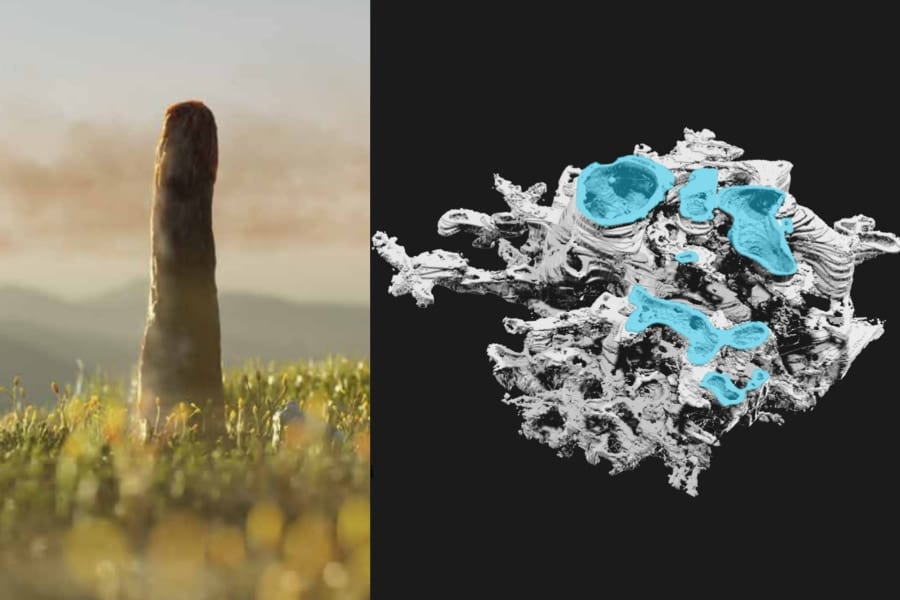
今回の研究で特に興味深いのは、“一瞬で壊れてしまうタンパク質”からでも、壊れる瞬間のデータを解析すれば細かな構造差までわかるかもしれない、という逆転の発想です。
たとえば、通常であれば「分子が壊れたら終わり」と考えがちです。
しかし、クーロン爆発の飛び散り方がきちんとしたパターンを示すなら、それを正しく読み取ることで、むしろ爆発前の姿—つまりタンパク質がどう折りたたまれていたか—を推定できる可能性があるわけです。
しかも今回、アミノ酸配列こそ同じでも、折りたたみ方や複合体の有無がほんの少し違うだけで爆発フットプリントが変化することが確認されました。
これは実験環境や分析手順をさらに工夫すれば、もっと微細な分子変化まで判別できるかもしれないという期待につながります。
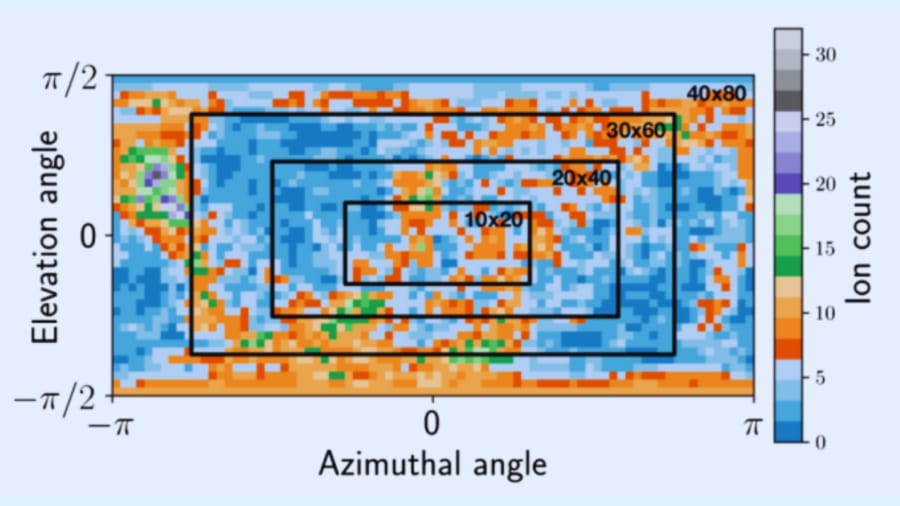
また、実際の実験に置きかえて考えると、ほんの一部分の角度しか検出器でカバーできなくても、意外と情報が失われずに識別できるというシミュレーション結果も示唆的です。
もしこの方式を本格的に適用できるようになれば、結晶化できないタンパク質の研究や、一時的な構造変化をとらえる研究で威力を発揮するかもしれません。
たとえばタンパク質が反応途中でどんなふうに形を変えるのか、爆発フットプリントの違いでリアルタイムに見分ける、といった応用も考えられそうです。
一方で、この研究はあくまでシミュレーションに基づいており、実際のXFELを使った実験でどこまで同じ精度が出せるか、タンパク質の向きをどう把握するか、といった課題は残ります。
ただ、最近は外部電場で分子の姿勢をある程度そろえる技術や、タンパク質内の特定元素(硫黄など)をタグのように利用する手法も提案されています。
そういった技術とクーロン爆発のパターン解析が組み合わされば、いずれは実験室レベルでも「爆発フットプリントだけで分子の全貌を推定する」という場面が来るかもしれません。
さらに、コンピュータでの構造予測ソフト(たとえばAlphaFoldなど)が進歩している今、実験結果を機械学習で解析して補完するのも有力です。
もし爆発フットプリントを何千、何万と集めれば、そのデータを学習させることで、未知のタンパク質がどんな立体構造を取りやすいか自動推定する…といった道も開けるでしょう。
つまり従来の「結晶化してX線回折を解析する」という手順とはまったく違う形で、生体分子の構造に挑む未来が見えてきます。
こうした点を踏まえると、今回の結果は単なる「爆発シミュレーションの成功」という枠を超えて、“壊れる瞬間”を活かすという新たな考え方を打ち出した、といえます。
結晶化という大きな壁、装置の制約、時間分解や配向などさまざまなハードルを乗り越える糸口として、クーロン爆発の解析が有望な選択肢となることを示唆しているのです。
今後、実験面での改良や理論的モデルの進化に伴い、この手法がどこまで一般的なツールになっていくのかが注目されます。
タンパク質を“爆発させる”という、一見奇抜な発想が、構造生物学の世界を大きく変えるかもしれません。


























![[W.P.S.] 防水スプレー 除菌 抗菌 防汚 機能付き 大容量420ml 日本製](https://m.media-amazon.com/images/I/41aHfy9uuZL._SL500_.jpg)
![[WILL CLENS] シューズパウダー 日本製 無香料 シューケアキット 靴 消臭 パウダー (1本)](https://m.media-amazon.com/images/I/41m8Ig2XigL._SL500_.jpg)
![よーく聞いてね!3つのヒントで学ぶ!どうぶつカード ([バラエティ])](https://m.media-amazon.com/images/I/51zT3OcliFL._SL500_.jpg)